Germany
March 22, 2021
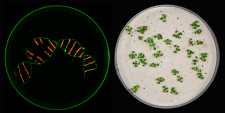
In their work, the researchers used markers to distinguish between different plant seeds. No difference can be seen with the naked eye. Under UV light, however, transgenic seeds appear red, non-transgenic seeds green. (left picture)
Foto: Jessica Lee Erickson
Using an improved version of the gene editing tool CRISPR/Cas9, researchers knocked out up to twelve genes in plants in a single blow. Until now, this had only been possible for single or small groups of genes. The approach was developed by researchers at Martin Luther University Halle-Wittenberg (MLU) and the Leibniz Institute of Plant Biochemistry (IPB). The method makes it easier to investigate the interaction of various genes. The study appeared in "The Plant Journal".
The inheritance of traits in plants is rarely as simple and straightforward as Gregor Mendel described. The monk, whose experiments in the 19th century on trait inheritance in peas laid the foundation of genetics, in fact got lucky. "In the traits that Mendel studied, the rule that only one gene determines a specific trait, for example the colour of the peas, happened to apply," says plant geneticist Dr Johannes Stuttmann from the Institute of Biology at MLU. According to the researcher, things are often much more complicated. Frequently there are different genes that, through their interaction with one another, result in certain traits or they are partly redundant, in other words they result in the same trait. In this case, when only one of these genes is switched off, the effects are not visible in the plants.
The scientists at MLU and IPB have now developed a way to study this complex phenomenon in a more targeted way by improving CRISPR/Cas9. These gene editing tools can be used to cut the DNA of organisms at specific sites. The team built on the work of biologist Dr Sylvestre Marillonnet who developed an optimised building block for the CRISPR/Cas9 system at the IPB. "This building block helps to produce significantly more Cas9 enzyme in the plants, which acts as a scissor for the genetic material," explains Stuttmann. The researchers added up to 24 different guide RNAs which guide the scissor enzyme to the desired locations in the genetic material. Experiments on thale cress (Arabidopsis thaliana) and the wild tobacco plant Nicotiana benthamiana proved that the approach works. Up to eight genes could be switched off simultaneously in the tobacco plants while, in the thale cress, up to twelve genes could be switched off in some cases. According to Stuttmann, this is a major progress: "As far as I know, our group has been the first to successfully address so many target genes at once. This may make it possible to overcome the redundancy of genes," says the biologist.
Until now, creating multiple mutations was a much more complex process. The plants had to be bred in stages with a single mutation each and then crossed with one another. "This is not only time-consuming, it’s also not possible in every case," says Stuttmann. The new approach developed at the MLU and the IPB overcomes these disadvantages and could prove to be a more efficient method of research. In future, it will also be possible to test random combinations of several genes in order to identify redundancies. Only in the case of conspicuous changes in the plant’s traits would it then be necessary to specifically analyse the genetic material of the new plants.
The study was supported by the Deutsche Forschungsgemeinschaft (DFG, German Research Foundation).
Study: Stuttmann J. et al. Highly efficient multiplex editing: One-shot generation of 8x Nicotiana benthamiana and 12x Arabidopsis mutants. The Plant Journal (2021). Doi: 10.1111/tpj.15197
Fortschritt für CRISPR/Cas: Forschende schalten viele Pflanzen-Gene auf einmal aus
Viele Mutationen auf einen Streich: Mit Hilfe einer verbesserten Variante der Gen-Schere CRISPR/Cas9 lassen sich bis zu zwölf Gene in Pflanzen auf einmal ausschalten. Bislang war das nur für einzelne Gene oder kleine Gruppen möglich. Entwickelt wurde der Ansatz von Forschenden der Martin-Luther-Universität Halle-Wittenberg (MLU) und des Leibniz-Instituts für Pflanzenbiochemie (IPB). Mit der Methode lässt sich das Zusammenspiel verschiedener Gene besser erforschen. Die Studie erschien in der Fachzeitschrift "The Plant Journal".
Die Vererbung von Eigenschaften ist bei Pflanzen selten so einfach und geradlinig wie von Gregor Mendel beschrieben. Der Mönch, der im 19. Jahrhundert mit seinen Versuchen zur Vererbung an Erbsen die Grundlagen der Genetik legte, hatte Glück: "Bei den von Mendel untersuchten Eigenschaften galt zufällig die Regel: Nur ein Gen bestimmt eine konkrete Eigenschaft, zum Beispiel die Farbe der Erbsen", sagt der Pflanzengenetiker Dr. Johannes Stuttmann vom Institut für Biologie der MLU. In vielen Fällen sei es aber deutlich komplizierter, so der Forscher: Oft sind es verschiedene Gene, die im Zusammenspiel in bestimmten Eigenschaften resultieren oder teilweise redundant zueinander sind, also dieselbe Eigenschaft zur Folge haben. Wird nur eins dieser Gene ausgeschaltet, sind die Folgen bei den Pflanzen nicht zu sehen.
Die Wissenschaftlerinnen und Wissenschaftler der MLU und des IPB haben nun eine Möglichkeit entwickelt, dieses komplexe Phänomen gezielter zu untersuchen. Dafür verbesserten sie die Gen-Schere CRISPR/Cas9, mit der sich die DNA von Organismen an bestimmten Stellen schneiden lässt. Grundlage hierfür war die Arbeit des Biologen Dr. Sylvestre Marillonnet, der am IPB einen optimierten Baustein für das CRISPR/Cas9-System entwickelte. "Mit Hilfe dieses Bausteins wird in den Pflanzen deutlich mehr von dem Cas9-Enzym gebildet, das als Schere für das Erbgut fungiert", sagt Stuttmann. Zusätzlich wurden bis zu 24 verschiedene sogenannte Guide-RNAs hinzugefügt, die das Scheren-Enzym an die gewünschten Stellen im Erbgut führen. Experimente an der Ackerschmalwand (Arabidopsis thaliana) und der wilden Tabakpflanze Nicotiana benthamiana zeigten, dass der Ansatz funktioniert: In den Tabakpflanzen konnten bis zu acht Gene, in der Ackerschmalwand in Einzelfällen sogar bis zu zwölf Gene gleichzeitig ausgeschaltet werden. Das sei ein großer Fortschritt, sagt Stuttmann: "Meines Wissens nach ist unsere Gruppe die erste, die so viele Zielgene auf einen Schlag erfolgreich ansprechen konnte. Damit lässt sich die Redundanz von Genen womöglich überkommen", so der Biologe.
Bislang waren die Möglichkeiten, solche Mehrfachmutationen zu erzeugen, deutlich aufwändiger: Die Pflanzen mussten mit jeweils einer einzelnen Mutation schrittweise gezüchtet und dann miteinander gekreuzt werden. "Das ist nicht nur zeitaufwändig, sondern auch nicht in allen Fällen möglich", sagt Stuttmann. Der neue, an der MLU und am IPB entwickelte Ansatz kompensiert diese Nachteile und stellt womöglich eine effizientere Methode für die Forschung dar: Künftig können so auch zufällige Kombinationen mehrere Gene ausprobiert werden, um Redundanzen aufzuzeigen. Erst bei auffälligen Veränderungen müsste dann gezielt das Erbgut der neuen Pflanzen analysiert werden.
Die Studie wurde von der Deutschen Forschungsgemeinschaft (DFG) gefördert.
Studie: Stuttmann J. et al. Highly efficient multiplex editing: One-shot generation of 8x Nicotiana benthamiana and 12x Arabidopsis mutants. The Plant Journal (2021). Doi: 10.1111/tpj.15197